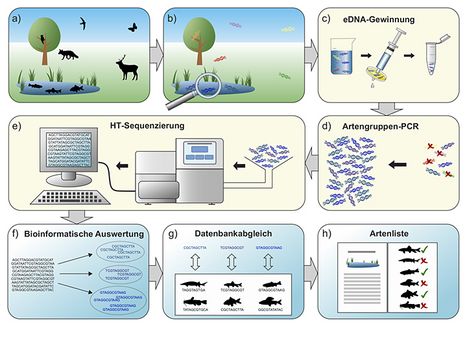
Workflow einer eDNA-basierten Erhebung am Beispiel einer Metabarcoding-Studie über Fische.
Aus Ausgabe 6/7-2023
Arterfassungen mittels Umwelt-DNA (eDNA) und die Bedeutung digitaler Sequenzinformationen für die Biodiversitätsforschung
Von Tamara Schenekar
Für Arterfassungen auf Basis von Umwelt-DNA (environmental DNA, eDNA) wird aus einer Umweltprobe DNA gewonnen und diese z. B. mittels Metabarcoding-Analyse sequenziert, wodurch mit minimaler Invasivität und sehr zeit- und kosteneffizient Informationen über die Anwesenheit ganzer Artengemeinschaften generiert werden können. Den vielen Vorteilen eDNA-basierter Erhebungen stehen allerdings noch einige offene Fragen gegenüber, die vor einer Implementierung dieser Ansätze in standardisierte Monitoringprogramme geklärt werden müssen, z. B. hinsichtlich der Aussagekraft in Bezug auf Abundanzen oder hinsichtlich der Dynamik von eDNA in der Umwelt. Nicht nur für Metabarcoding-Analysen sind digitale Sequenzinformationen eine wichtige Datengrundlage in Naturschutz und Biodiversitätsforschung. Öffentlich zugängliche Sequenzdatenbanken wie GenBank und das Barcode of Life Data System (BOLD) erlauben der Wissenschaft rasante Fortschritte in vielen Forschungsbereichen. Allerdings steht der offene Zugang zu generierten Sequenzdaten evtl. im Konflikt mit dem Nagoya-Protokoll, das die faire Nutzung genetischer Ressourcen und einen gerechten Vorteilsausgleich zwischen den Vertragsstaaten regelt. Hier sind klare und einfache Lösungen erforderlich, um digitale Sequenzinformationen fair und effizient für Forschung und Biodiversitätsschutz einsetzen zu können.
DOI: 10.19217/NuL2023-06-02