Thomas Borsch und Elke Zippel
Zusammenfassung
Angesichts des derzeitigen drastischen Verlusts pflanzlicher Vielfalt bekommt das Fachgebiet der Naturschutzgenetik weltweit wachsende Aufmerksamkeit. Die technischen Fortschritte, mit denen wir durch die Entschlüsselung der Genome von Organismen Einblicke in deren Evolution erhalten, können auch für den Schutz der heimischen Flora gewinnbringend genutzt werden. Für den Naturschutz, der zum Ziel hat, die genetische Integrität von Arten und deren Evolutionspotenzial in der sich verändernden Landschaft zu erhalten, haben molekulargenetische Daten eine herausragende Bedeutung. Mit Hilfe solcher naturschutzgenetischer Informationen können Populationsstützungen und Wiederansiedlungen gefährdeter Arten gezielt entwickelt werden. Genauso bildet eine Kenntnis der innerartlichen genetischen Vielfalt die Grundlage für den Einsatz von Regiosaatgut. Bisher liegen allerdings nur von wenigen mitteleuropäischen Arten entsprechende ausführliche Daten über ihr gesamtdeutsches
Verbreitungsgebiet vor. Diese zeigen stets artspezifische Muster, die die dynamische Evolutionsgeschichte der Arten bis zu rezenter Fragmentierung von Populationen widerspiegeln. Um das Potenzial der Naturschutzgenetik in Zukunft besser und v. a. zielgerichtet einsetzen zu können, ist eine starke Vernetzung von Akteuren aus praktischem Naturschutz und Wissenschaft notwendig. Wir regen an, bundesweit geeignete Strukturen und Programme zu schaffen sowie einen regelmäßigen Austausch und den Aufbau von Kooperationen zu fördern.
Naturschutzgenetik – genetische Vielfalt – genetisches Monitoring – Vernetzung von Naturschutz und Wissenschaft – mitteleuropäische Flora – molekulare Techniken – DNA-SequenzierungAbstract
The field of conservation genetics is gaining recognition in light of the current dramatic losses of plant diversity. Rapid methodological and technological advances in uncovering genomic information of organisms not only make it possible to illuminate their evolutionary history but can also be applied successfully to native plant conservation. Practical conservation measures that aim at maintaining the evolutionary potential of species in changing landscapes can benefit enormously from molecular genetic information. Genetic data support the design of effective species conservation strategies. For those species that have been investigated throughout their ranges in Germany, species-specific patterns of genetic differentiation of their populations as well as genetic variation within populations are evident. Patterns of genetic diversity can be explained by the evolutionary histories of species but can also reflect the impacts of recent decline and isolation.
Genetic data already serve conservation management, including regional seed sourcing and the recovery and re-introduction of populations of endangered species. In order to fully exploit the potential offered by conservation genetics, a much stronger interaction between stakeholders is needed, encompassing applied and scientific fields. This could be achieved by establishing firm networks and programmes integrating federal and regional to state levels, and by promoting regular knowledge exchange and supporting collaborative activities.
Conservation genetics – Genetic diversity – Genetic monitoring – Networking of conservation and science – Central European flora – Molecular techniques – DNA sequencingInhalt
1 Einleitung
Die drei Ebenen der biologischen Vielfalt (innerartliche Vielfalt, Artenvielfalt und Habitatvielfalt) werden durch das Übereinkommen über die biologische Vielfalt (Convention on Biological Diversity, CBD) explizit anerkannt. Die Nationale Strategie zur biologischen Vielfalt als Umsetzung der CBD in Deutschland entspricht der Bedeutung dieser drei Ebenen mit dem ausdrücklichen Ziel, „eine Vielfalt von regional angepassten Populationen zu erhalten und den natürlichen genetischen Austausch wildlebender Arten sicherzustellen“ (BMU 2007: Kapitel B 1.1.4).
Die innerartliche genetische Vielfalt wird von verschiedenen Faktoren geprägt und zeigt artspezifische Muster, die zunächst ein Ergebnis von Evolutionsprozessen in Raum und Zeit sind. Neben den weit verbreiteten Artbildungsmechanismen durch räumliche Isolation (allopatrische Artbildung) spielen bei Blütenpflanzen Hybridisierung und Polyploidie eine wichtige Rolle (Rieseberg, Willis 2007; Weiss-Schneeweiss et al. 2013). Bei der Allopolyploidisierung entstehen aus Vorfahren unterschiedlicher Arten fertile Nachkommen mit verdoppelten Chromosomensätzen und als Ergebnis neue Arten – und dies oft sogar mehrfach unabhängig voneinander. Für die Evolution der mitteleuropäischen Flora spielte dabei die klimatische Entwicklung im Quartär eine große Rolle, die im Wechsel von Eis- und Warmzeiten zu Wanderungen von Pflanzen geführt hat (z. B. Taberlet et al. 1998; Harter et al. 2015; Leipold et al. 2017), verbunden mit der Entstehung und Ausbreitung neuer Genotypen oder auch der Reduktion von Arealen und der Akkumulation unterschiedlicher Genotypen in kleinen Refugialräumen (Comes, Kadereit 1998). Diese Dynamik in Zeit und Raum hat einerseits immer wieder zu Kreuzungen und Rückkreuzungen mit Austausch von Genen zwischen unterschiedlichen Populationen und Arten geführt (Introgressionen) und andererseits bedingt, dass nicht alle Genvarianten (= Allele) oder Haplotypen einer Abstammungslinie auch konsistent in den Genomen einzelner Arten auftreten (bekannt als incomplete lineage sorting, Avise 2000). Um Daten zur genetischen Vielfalt von Arten richtig einordnen zu können, müssen solche Aspekte ihrer
Evolutionsgeschichte bekannt sein. Zusätzlich wird das spezifische Evolutionsgeschehen insbesondere von unterschiedlichen Reproduktions- und Ausbreitungssystemen der Arten beeinflusst, die eine wesentliche Rolle beim genetischen Austausch zwischen Individuen und Populationen spielen (Barrett 2010). All dies führt zu komplexen Mustern genetischer Vielfalt, die als Ausdruck der Evolutionsgeschichte von Arten im Rahmen der letzten Jahrtausende bis Jahrmillionen zu verstehen sind. Dazu gehören auch lokale Anpassungen von Genotypen an spezifische Umweltfaktoren (Leimu, Fischer 2008), die wiederum Auswirkungen auf funktionale Eigenschaften und Fitness (Montalvo, Ellstrand 2001) haben.
Mit zunehmender Gestaltung der Landschaft durch den Menschen wurde diese natürliche Dynamik v. a. bei zahlreichen Offenlandarten, die sich an den neuen Standorten auf Äckern, Wiesen und Weiden ausbreiten konnten (Poschlod 2015), verstärkt, was bis zur Ausbildung neuer Ökotypen in Anpassung an veränderte Standort- und Nutzungsbedingungen, wie etwa die regelmäßige Mahd, führte. Mit der Intensivierung der Landwirtschaft wird dieser Trend wieder umgekehrt. Die zunehmende Fragmentierung der Areale von Pflanzenarten verändert die genetische Struktur von Populationen (z. B. van Geert et al. 2008), wobei sich die historische Entwicklung der Landnutzung bis hin zur heutigen Landschaftsstruktur auf die genetische Vielfalt von Pflanzenarten auswirkt (Pagel et al. 2020). Das betrifft insbesondere einst weit verbreitete
Arten der Trockenrasen, Wiesen und Niedermoore (Wesche et al. 2012) oder anderer nährstoffarmer Habitate (Metzing et al. 2018). Folgen der räumlichen Isolation sind Inzucht in kleinen Populationen und die Tatsache, dass genetische Drift, also der stochastische Verlust von Allelen, nicht mehr durch Genfluss ausgeglichen werden kann (Oostermeijer et al. 2003; Aguilar et al. 2008). Solche genetischen Erosionsprozesse führen dann oft zu reduzierter Fitness und vermindern die Überlebenswahrscheinlichkeit der Bestände. Gefährdete Arten können daher trotz einer nach Artenschutzkriterien ausgerichteten Biotoppflege und Unterschutzstellung der verbliebenen isolierten Populationen verschwinden (Reichel et al. 2016).
Vor dem Hintergrund der historischen Dynamik und des globalen Wandels setzt sich zunehmend die Erkenntnis durch, dass Artenschutz heute nicht Arten als simple Einheiten konservieren kann, sondern vielmehr die Überlebensfähigkeit von Arten in einer veränderten und sich verändernden Landschaft im Blick haben muss. Es gilt also, das evolutive Potenzial von Arten unter sich verändernden Umweltbedingungen (Grivet et al. 2008) zu sichern. Die Erfassung der räumlichen Verteilung der genetischen Variabilität einer Art liefert hierbei die Grundlage, um ein repräsentatives Spektrum an Genotypen zu erhalten. Dabei kann die Erkennung von Refugialpopulationen auch außerhalb der klassischen europäischen Refugialgebiete (z. B. Harter et al. 2015) von genauso hoher Bedeutung sein wie die Identifikation seltener Genotypen an Arealrändern (Lesica,
Allendorf 1995), wo sich Populationen oft durch spezifische Anpassungen auszeichnen.
Diese Muster und die Dynamik der genetischen Ebene biologischer Vielfalt lassen sich heute gut detektieren, weil sich die DNA-Sequenzierung sowie evolutionsbiologische bzw. genomische Methoden rasant entwickeln. Damit werden genetische Daten zunehmend für die konkrete Umsetzung regionaler und lokaler Artenschutzmaßnahmen zugänglich. Molekulare Analysen können klären, ob sich eine ggf. vorhandene Fragmentierung und Isolation auf die genetische Struktur und Fitness der verbliebenen Populationen auswirken und ob bereits genetische Erosion vorliegt. Und sie bringen Hinweise dafür, wie groß Populationen einer Art mindestens sein müssen und welche Rolle im konkreten Fall der Genfluss für die Erhaltung der Fitness ihrer Populationen spielt. Daraus kann beurteilt werden, ob Ansiedlungen zur Ermöglichung von Genfluss oder Populationsstützungen mit genetischem Material anderer Populationen sinnvoll sind und, wenn ja, welche Populationen sich als Materialquelle eignen (genetic
rescue).
Molekulargenetische Untersuchungen sind auch vonnöten, um mögliche Introgressionen zu detektieren, die von Kultursorten oder gebietsfremden Zierpflanzen ausgehen und den Genpool heimischer Arten verändern. So zeigten Nierbauer et al. (2014), dass Nymphaea alba (Weiße Seerose) in der Mehrzahl hessischer Fundorte aus Hybriden mit Kultursorten der nordamerikanischen Nymphaea odorata (Wohlriechende Seerose) besteht. Hybridisierung mit Zierpflanzen ist damit ein großes Problem für den Fortbestand der gefährdeten einheimischen Weißen Seerose. Andererseits konnten etwa Reim et al. (2017) mittels molekularer Marker klären, welche Bestände reine Populationen von Pyrus pyraster (Wildbirne) sind, um sie in Artenschutzprogramme aufzunehmen.
Voraussetzung für die Analyse der genetischen Vielfalt von Arten ist, dass Artgrenzen nach heutigen wissenschaftlichen Maßstäben verstanden sind (Naciri, Linder 2015) und Taxonkonzepte auf phylogenetisch-evolutionsbiologischen Erkenntnissen beruhen. Denn nur so ist gewährleistet, dass die etwa in Checklisten akzeptierten Arten auch wirkliche biologische Einheiten darstellen und damit Populationen, die im Rahmen von Artenschutzmaßnahmen, bei Habitatrestaurierungen/-renaturierungen (mit Regiosaatgut) oder bei der Nutzung genetischer Ressourcen eine Rolle spielen, im richtigen Kontext bearbeitet werden. Die rasante methodische Entwicklung bedingt aber auch, dass wir uns in einer Umbruchphase von der Alpha-Taxonomie hin zu evolutionsbiologisch-phylogenetisch fundierten Taxonkonzepten befinden. Populationsgenetische und phylogeographische Ansätze sind daher auch Teil der Analyse von Artgrenzen. Hier erfordert moderne Forschung
zur Erfassung der Artenvielfalt internationales Zusammenwirken und dynamische Informationssysteme, die neue wissenschaftliche Erkenntnisse kontinuierlich und länderübergreifend einpflegen und als taxonomische Referenzsysteme verfügbar machen (Borsch et al. 2020). Angesichts der Dynamik der Entstehung der mitteleuropäischen Flora betrifft dies Sippenkomplexe in vielen Gattungen der Blütenpflanzen. So weist die Liste der Gefäßpflanzen Deutschlands (Buttler et al. 2018) durch zahlreiche sog. „Aggregate“ auf kritische Sippenkomplexe hin.
Auch wenn die Berücksichtigung populationsgenetischer Parameter bei gezielten Artenschutzmaßnahmen langsam steigt (Mijangos et al. 2014), gehen im praktischen Naturschutz, wie etwa bei Ansiedlungen oder Populationsstützungen, oft keine genetischen Analysen voraus (Godefroid et al. 2011; Shafer et al. 2014). Trotz der hohen Relevanz für den Artenschutz entzieht sich molekulare und populationsgenetische Forschung vielfach methodisch und technisch dem Erfahrungsbereich von Akteuren des praktischen Naturschutzes. In Kooperation mit wissenschaftlichen Einrichtungen ergibt sich jedoch eine Reihe von Möglichkeiten, Ergebnisse der Naturschutzgenetik stärker zu berücksichtigen. In diesem Beitrag geben wir anhand einiger Beispiele aus Deutschland einen Einblick in den Stand der Forschung und skizzieren Perspektiven und
Erfordernisse für die Zukunft.
2 Genetische Vielfalt von Arten der einheimischen mitteleuropäischen Flora
2.1 Weiträumige Muster innerartlicher genetischer Vielfalt
Die genetische Vielfalt innerhalb von Populationen und die genetische Differenzierung zwischen Populationen sind erst bei wenigen Blütenpflanzenarten repräsentativ über deren gesamtes Verbreitungsgebiet in Deutschland untersucht. Ein Großteil der publizierten Arbeiten beschäftigt sich mit Hochgebirgsarten der Alpen, meist mit phylogeographischen Fragestellungen (z. B. Albach et al. 2006). Phylogeographische Untersuchungen von Steppenpflanzen wie Stipa-Arten (Federgräser; Hensen et al. 2010; Durka et al. 2013), Scorzonera purpurea (Violette Schwarzwurzel; Meindl et al. 2016) oder Adonis vernalis (Frühlings-Adonisröschen; Kropf et al. 2020) zeigten eine klare genetische Differenzierung der
deutschen Populationen gegenüber den Populationen im größeren östlichen und südöstlichen Verbreitungsgebiet. Andere Autoren widmeten sich der genetischen Variation innerhalb von Populationen in Abhängigkeit von historischen und ökologischen Faktoren. So fanden Reisch, Rosbakh (2021) in einer Metaanalyse, dass die genetische Differenzierung von Populationen des Tieflands hin zu höher gelegenen Populationen der alpinen Zone anstieg, während im Gegensatz dazu die genetische Vielfalt innerhalb alpiner Populationen deutlich geringer war.
Huck et al. (2012) konnten bei der in Bergwiesen einiger Mittelgebirge typischen Art Meum athamanticum (Bärwurz) refugiale Muster finden. Durka et al. (2017) haben Herkünfte einiger weit verbreiteter Wiesenpflanzen wie Arrhenatherum elatius (Glatthafer), Knautia arvensis (Wiesen-Witwenblume) oder Lychnis flos-cuculi (Kuckucks-Lichtnelke) aus verschiedenen Herkunftsregionen für Regiosaatgut (Prasse et al. 2010) untersucht und dabei eine deutliche genetische Differenzierung festgestellt. Die genetischen Marker ergaben je nach Art unterschiedlich starke Evidenz für räumliche Isolation und adaptive Prozesse (Durka et al. 2017). Allerdings hat das Sampling-Design solcher Untersuchungen Einfluss auf die Ergebnisse. So konnten Listl et al. (2017) an bayerischen Populationen von Lathyrus pratensis (Wiesen-Platterbse) feststellen, dass die genetische Variabilität und seltene Allele mit größerer Nähe zu den Alpen zunehmen und die innerartlichen Muster genetischer Vielfalt damit komplexer sind als das Raster naturräumlich definierter Herkunftsregionen. Daten liegen außerdem für einige Gehölze vor, z. B. für Prunus spinosa (Schlehe; Eimert et al. 2016). Hier wurden eine mäßige genetische Differenzierung in zwei große geographische Cluster sowie Gründereffekte ausgehend von unterschiedlichen Erstbesiedlungsereignissen festgestellt. Der überwiegende Teil der vorliegenden Studien beruht auf amplified fragment length polymorphisms (AFLP; Kasten 1) als Markersystem. Dazu kamen Sequenzen aus Regionen des Plastidengenoms und der ribosomalen
DNA des Kerngenoms (internal transcribed spacers, ITS) bei phylogeographischen Analysen.
Kasten 1: Molekulare Werkzeuge in der Naturschutzgenetik.
Box 1: Molecular tools in conservation genetics.
In den letzten Jahrzehnten wurden verschiedene molekularbiologische Techniken mit entsprechenden Typen genetischer Marker entwickelt, die für die Bearbeitung naturschutzgenetischer Fragestellungen eingesetzt werden können. Hinsichtlich der verschiedenen Fragestellungen, die sich mit Hilfe der unterschiedlichen Markersysteme bearbeiten lassen, sei weiterführend auf Holderegger, Segelbacher (2016) verwiesen.
Sequenzen von Genomregionen: Pflanzen haben drei genomische Kompartimente, das Kerngenom im Zellkern sowie die sehr viel kleineren Organellengenome der Chloroplasten und Mitochondrien. Genomregionen sind dabei strukturell und funktionell klar definierte Abschnitte (z. B. Gene, Introns, Spacer). Vergleicht man Sequenzen homologer (auf einen gemeinsamen Vorfahren zurückgehender) Genomregionen bei unterschiedlichen Individuen von Populationen oder Arten, kann eine mehr oder weniger große Zahl von Mutationen gefunden werden. Solche Unterschiede in DNA-Merkmalen erlauben die Rekonstruktion von Stammbäumen und oft auch der großräumigen geographischen Differenzierung von Arten (Phylogeographie). Die Regionen des Plastidengenoms und der ribosomalen DNA des Kerngenoms (dazu gehören die häufig sequenzierten internal transcribed spacer, ITS) zeigen jedoch zu wenig innerartliche Variabilität und eignen sich deshalb nicht für populationsgenetische
Studien.
Mikrosatelliten sind Abschnitte mit Wiederholungen von einem bis wenigen Basenpaaren in klar definierten Genomregionen (Loci). Sie sind hochvariabel, und bei der Analyse können alle vorhandenen Allele der jeweiligen Loci identifiziert sowie homo- und heterozygote Genotypen unterschieden werden. Dies bietet für populations- und naturschutzgenetische Fragestellungen entscheidende Vorteile. Es ist recht aufwendig, die jeweils in der Regel artspezifischen Mikrosatelliten-Loci im Genom zu finden. Wurde diese Markerentwicklung einmal durchgeführt, hat die Methode jedoch den Vorteil, dass sie leicht für eine große Zahl von Proben eingesetzt werden kann und die Daten sehr gut reproduzierbar sind. Datensätze, die mit den gleichen Mikrosatelliten-Loci in verschiedenen Laboren zu verschiedenen Zeiten generiert wurden, können beliebig erweitert werden.
Single nucleotide polymorphisms (SNP) sind Stellen im Genom, an denen sich zwei Individuen durch ein unterschiedliches Basenpaar voneinander unterscheiden. Ähnlich wie Mikrosatelliten sind sie gut reproduzierbar, müssen aber durch eine vorausgehende Analyse des Genoms charakterisiert werden. Interessant ist, dass mit ihnen anpassungsrelevante Teile des Genoms und damit funktionelle genetische Vielfalt (adaptive genetische Vielfalt) erfasst werden kann. Dies ist durch High-throughput-Sequenziermethoden möglich, bei denen sehr große Mengen kurzer Sequenzen generiert werden. Die Variabilität potenzieller SNP-Positionen ist sehr verschieden, daher müssen SNP den Fragestellungen entsprechend angepasst werden.
Amplified fragment length polymorphism (AFLP): Mit Hilfe von Enzymen wird die Gesamt-DNA an definierten Basenfolgen geschnitten und in Folgeschritten ein Teil dieser unterschiedlich langen DNA-Stränge mittels einer Polymerase-Kettenreaktion amplifiziert. Nach der Elektrophorese erhält man Bandenmuster, die jeweils für Individuen, Populationen bzw. Cluster verschiedener Populationen charakteristisch sind. Ein genetisches Fingerprinting ist vergleichsweise einfach mit AFLP durchzuführen. Die Methode hat aber den Nachteil, dass die Reproduzierbarkeit eingeschränkt ist und Datensätze kaum erweiterbar sind. Daher werden AFLP zunehmend von Mikrosatelliten und SNP abgelöst.
In den letzten Jahren wurden für deutschlandweit verbreitete gefährdete Arten mit Hilfe von Hochdurchsatzsequenziermethoden Kernmikrosatellitenmarker entwickelt, so für Arnica montana (Arnika; Duwe et al. 2015), Crepis mollis (Weicher Pippau; Duwe et al. 2016) und Scabiosa canescens (Graue Skabiose; Ismail et al. 2015). Diese Forschungen gehören zu einem Programm am Botanischen Garten und Botanischen Museum Berlin (BGBM), in dem Populationen gefährdeter Blütenpflanzen aus ganz Deutschland vergleichend populationsgenetisch untersucht werden, um sowohl evolutionsgeschichtlich bedingte Muster innerartlicher Differenzierung aufzuklären als auch die Vielfalt eines repräsentativen Spektrums von Populationen besser zu verstehen. Die drei zuerst ausgewählten Arten stehen stellvertretend für Pflanzen extensiv
bewirtschafteter Grünlandhabitate, deren Populationen durch Landnutzungswandel, Fragmentierung und teils drastische Verkleinerung bis hin zum Erlöschen besonders betroffen sind. Die Arbeiten wurden im Umweltforschungsplan (Bundesamt für Naturschutz, BfN) gefördert. Für Arnica montana sind in Abb. 1 die Ergebnisse aus dem gesamten deutschen Verbreitungsgebiet basierend auf Duwe et al. (2017) mit Bezug zur Bestandsentwicklung anhand der floristischen Kartierung dargestellt. Die Populationen zeigen dabei neben einem breiten Spektrum der genetischen Vielfalt innerhalb der untersuchten Populationen eine außerordentlich starke geographische Differenzierung (Fixierungsindex FST = 0,27) zwischen den Populationen mit zahlreichen jeweils einzigartigen (sog. „privaten“) Allelen. Um die geographischen Muster noch besser zu verstehen, ist es wichtig, weitere
Populationen zu beproben. Dazu haben sich Forscher auf bestimmte Mikrosatelliten-Loci geeinigt, so dass Datensätze verschiedener Untersuchungen integrierbar werden. Eine solche Zusammenarbeit findet z. B. bei den für Arnica entwickelten Mikrosatelliten-Loci schon statt (Titze et al. 2020; S. Liepelt, persönliche Kommunikation). Auch bei Scabiosa canescens, einer konkurrenzschwachen Art basenreicher Halbtrocken- und Sandtrockenrasen in vorwiegend wärmeren Regionen Deutschlands, konnten Ismail et al. (2018) anhand von 22 Populationen zeigen, dass sich die Genotypen dieser Art in verschiedenen Naturräumen Deutschlands deutlich voneinander unterscheiden und hier v. a. die Populationen im Rheintal stark abweichen. Demgegenüber zeigte Crepis mollis im deutschen Teilareal von den Mittelgebirgen bis in die Alpen eine nur mäßige
genetische Differenzierung, aber drei genetische Cluster (1. Harz, Erzgebirge und Thüringer Wald; 2. Vogelsberg und Sauerland; 3. Alpenvorland und Alpen).
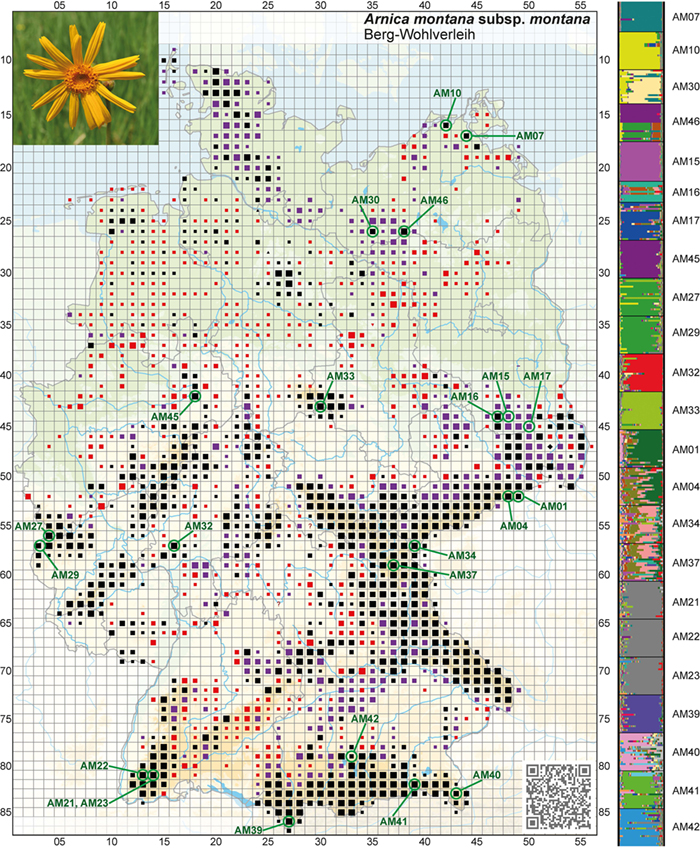
Abb. 1: Visualisierung der genetischen Differenzierung von Arnica montana (Arnika, Berg-Wohlverleih) basierend auf einer Bayes-Clusteranalyse (STRUCTURE-Analyse, K = 24) und Daten von Kernmikrosatelliten der untersuchten deutschen Populationen (nach Duwe et al. 2017, verändert). Die unterschiedlichen Farben (rechts) beziehen sich auf die verschiedenen gefundenen Gruppen von Genotypen: Jede Farbe repräsentiert einen Cluster von Genotypen. Die untersuchten Populationen (grün umrandet) sind dabei im Kontext der floristischen Daten in FloraWeb (Bundesamt für Naturschutz, https://www.floraweb.de/) dargestellt. Schwarze Symbole stehen für einen Nachweis nach 1980 (Kartengrundlage: NetPhyD/BfN 2013).
Fig. 1: Visualisation of the genetic differentiation of
Arnica montana (arnica) based on a Bayesian Clustering analysis (STRUCTURE analysis, K = 24) and data of nuclear microsatellites from the German populations investigated (based on
Duwe et al. 2017, modified). The different colours (to the right) correspond to the encountered different groups of genotypes: each colour represents a cluster of genotypes. The populations investigated (with green circles) are depicted in relation to floristic data in FloraWeb (Federal Agency for Nature Conservation,
https://www.floraweb.de/). Black symbols indicate the presence of the species after 1980 (map basis:
NetPhyD/BfN 2013).
Die bisherigen Ergebnisse zur genetischen Vielfalt von Blütenpflanzen in Deutschland legen nahe, dass Arten trotz ähnlicher Verbreitungsgebiete und vergleichbarer Habitate kaum korrespondierende räumliche Muster zeigen. Dies betrifft insbesondere die geographische Differenzierung der Populationen. Dafür sind offensichtlich die artspezifischen Evolutionshistorien zu verschieden. Auch die Unterschiede in den Reproduktionssystemen der Arten sind zu groß, wie auch aus den Niederlanden bekannt ist (Oostermeijer et al. 2003). Grundsätzlich ist aber festzuhalten, dass auch bei Arten seminatürlicher Lebensräume wie Grünlandgesellschaften eine deutliche geographische Differenzierung der innerartlichen genetischen Vielfalt vorliegt, was auf autochthone, regional unterschiedliche Genpools hinweist.
2.2 Naturschutzgenetische Erkenntnisse zu Populationen gefährdeter Arten
Von besonderer Bedeutung sind seltene und gefährdete Arten, die ihren Verbreitungsschwerpunkt ausschließlich in Mitteleuropa haben und für deren Schutz Deutschland eine hohe Verantwortung trägt (Welk 2002; Ludwig et al. 2007). Der größte Teil dieser Pflanzen gehört zu den sog. Verantwortungsarten (Gruttke et al. 2004), für deren Schutz eigens ein Schwerpunkt im Bundesprogramm Biologische Vielfalt (BPBV) eingerichtet wurde. Da sie im besonderen Fokus des Artenschutzes stehen, sollten sie auch besondere Aufmerksamkeit in der naturschutzgenetischen Forschung erfahren.
Spezifische naturschutzgenetische Fragestellungen wurden bisher für einige kleinräumig verbreitete Arten bearbeitet: So hat Becker (2003) dargelegt, dass kleine Populationen von Astragalus exscapus (Stängelloser Tragant) in Deutschland durch Drift und Inzucht ihre genetische Variabilität verloren und Fitness eingebüßt haben, aber dennoch seltene Allele aufweisen. Die Erweiterung kleinräumiger Nischen zur besseren Etablierung von Jungpflanzen in Nachbarschaft der Mutterpflanzen sowie die gezielte Zuführung genetischen Materials über Samen aus gut bekannten Populationen derselben Teilareale wurden empfohlen. Im Gegensatz dazu wurde bei der natürlich auf isolierten Kalkfelsen vorkommenden Pfingst-Nelke (Dianthus gratianopolitanus) bei stärkerem Isolationsgrad der Populationen keine genetische Erosion gefunden (Putz et al. 2015). Die genetische Variabilität nahm bei
größerer Populationsdichte sogar ab.
Mit Blick auf den Status der Populationen der deutschlandweit verbreiteten Arnica montana fanden Duwe et al. (2017) v. a. bei den Restvorkommen im norddeutschen Tiefland genetische Erosion. Die Art ist außerhalb der Alpen und in Teilen der Mittelgebirge in den letzten Jahrzehnten stark zurückgegangen (Abb. 1). Da die Tieflandpopulationen sich genetisch deutlich von den Populationen der Mittelgebirge und der Alpen unterscheiden, sind die Tieflandgenotypen und damit ein bestimmter Teil der genetischen Vielfalt der Art besonders gefährdet. Bei Scabiosa canescens weisen die Daten von Ismail et al. (2018) darauf hin, dass früher vorhandener Genfluss innerhalb von Naturräumen heute nicht mehr existiert. Bei beiden Arten ist es wahrscheinlich, dass manche Populationen selbst bei angepasster Habitatpflege
bzw. Wiederherstellung geeigneter Standortbedingungen auf Grund ihrer genetischen Verarmung nicht mehr dauerhaft überlebensfähig sind. Hier sind gezielte Maßnahmen der Bestandsstützung und genetischen Auffrischung sinnvoll.
Malus sylvestris, der einheimische Wildapfel, gehört zwar nicht zu den Verantwortungsarten, ist aber ein seltenes Gehölz mit einem Vorwarnstatus in der Roten Liste (Metzing et al. 2018). Ein bundesweites Screening basierend auf Mikrosatelliten im Rahmen forst-genetischer Programme zur Erhaltung genetischer Ressourcen ergab eine räumliche genetische Differenzierung in fünf Großgruppen von Genotypen, wobei aber einige Populationen eine Hybridisierung mit genetisch deutlich verschiedenen Kulturäpfeln zentralasiatischen Ursprungs zeigten (Höltken et al. 2017). Die autochthonen Genotypen von Malus sylvestris und die genetische Integrität der Art sind daher besonders gefährdet.
3 Konsequenzen für den botanischen Artenschutz
Auf Grund der aktuellen Bestandssituation vieler Arten in der intensiv genutzten Landschaft Mitteleuropas ist ein zwischen Praxis und Wissenschaft eng abgestimmtes Handeln dringend geboten. Molekulargenetische Untersuchungen verdeutlichen, dass für einen wirksamen Artenschutz sowohl die genetische Integrität als auch die innerartliche genetische Vielfalt der Arten beachtet werden muss. Für die Förderung eines solchen wissenschaftlich fundierten Artenschutzes gibt es nach unserer Ansicht eine Reihe von Möglichkeiten und Prioritäten, die wir hier skizzieren.
3.1 Relevanz genetischer Erkenntnisse für die Praxis
Die Anwendung wissenschaftlicher Ergebnisse in der Praxis lässt sich an verschiedenen Themenbereichen konkreter illustrieren. Die wichtigsten Handlungsfelder sind:
1. Prioritätensetzung bei direkten Schutzmaßnahmen in situ. Artspezifische genetische Daten erlauben dabei Aussagen, welche Populationen bzw. Arealteile besonders wichtig sind, um die innerartliche Vielfalt repräsentativ zu erhalten. Ebenso lässt sich ableiten, wo Refugien mit vielen Genotypen oder Populationen mit seltenen Allelen liegen. Dieselben Daten können genauso die Planung von Ex-situ-Maßnahmen für die jeweiligen Zielarten unterstützen.
2. Analysen der genetischen Vielfalt und Struktur konkreter Populationen lassen erkennen, wo genetische Erosion vorliegt und spezifische Maßnahmen ergriffen werden sollten (assistierter Genfluss) und wo nicht. Kleine Populationen sind nicht per se genetisch verarmt. Mit populationsgenetischen und -biologischen Untersuchungen kann abgeschätzt werden, welche Mindestgröße eine überlebensfähige Population haben sollte, ab der keine Gefahr einer genetischen Drift und Inzucht besteht.
3. Genetische Daten sind insbesondere für die Planung, Begleitung und Evaluierung von Wiederansiedlungsmaßnahmen und Populationsstützungen wünschenswert (Betz et al. 2013; van Rossum et al. 2020) und in manchen Fällen auch erforderlich (Engelhardt et al. 2014), um für die Maßnahmen geeignetes Ursprungsmaterial zuverlässig zu identifizieren. Da eine zu geringe genetische Vielfalt die Überlebensfähigkeit von Populationen und Arten negativ beeinflussen kann, ist es in der Regel sinnvoll, das in einer Region vorhandene Material zur Erhöhung der Vielfalt zu mischen (z. B. Godefroid et al. 2011). Andererseits kann es durch Vermischen und anschließenden Genfluss zwischen zu distanten und unterschiedlich angepassten Genotypen selten auch zum Zusammenbruch
koadaptierter Genkomplexe und zu reduzierter Fitness kommen (Auszuchtdepression; Keller et al. 2000). 4. Bei Arten, die mit gebietsfremden exotischen Arten oder Kultursorten hybridisieren, kann der Genpool überflutet werden und in der Folge die Art aussterben (Holderegger, Segelbacher 2016); genetische Daten sind hier grundsätzlich erforderlich, um geeignetes Material für In-situ- und Ex-situ-Maßnahmen zu identifizieren.
Die Übertragung der Ergebnisse konkreter genetischer Studien in die Naturschutzpraxis ist bisher noch selten (Mijangos et al. 2014), nimmt aber zu. Ein Beispiel ist die Planung der Populationsstützung und Wiederansiedlung des Endemiten Cochlearia bavarica (Bayerisches Löffelkraut), für die alle verbliebenen 32 Populationen genetisch untersucht wurden (Kaulfuß, Reisch 2017). Die Ergebnisse von Ismail et al. (2018) werden für Ansiedlungen und Populationsstützungen von Scabiosa canescens in Berlin und Brandenburg im Rahmen des Projekts „Wildpflanzenschutz Deutschland“ (WIPs-De; Lauterbach et al. 2019) genutzt. Vermehrt werden genetische Daten bereits für die Erkennung gebietseigener und artreiner genetischer Ressourcen von Gehölzen eingesetzt, deren Genpools
potenziell von Hybridisierung mit exotischen Arten oder Kultursorten betroffen sind, so bei Malus sylvestris (Höltken et al. 2017) oder Populus nigra (Schwarzpappel; Jelić et al. 2015). Durch die Forstgenetik ist der Einsatz genetischer Daten bei Waldbäumen traditionell stärker etabliert. Dabei geht es z. B. um die Frage, welche Auswirkungen Saatguterntestrategien auf die genetische Vielfalt der neu begründeten Bestände haben (Gefahr von Flaschenhalseffekten und letztlich menschengemachter Gendrift) oder wie sich Umweltveränderungen auf die genetische Vielfalt auswirken (z. B. Schmiedel et al. 2017).
Einen pragmatischen Ansatz zur Berücksichtigung innerartlicher genetischer Differenzierung verfolgt das Regiosaatgutkonzept, das mit Blick auf die Saatgutproduktion für Begrünungsmaßnahmen Ursprungsgebiete auf geographisch-naturräumlicher Ebene definiert (z. B. Prasse et al. 2010). Die schon jetzt vorliegenden Ergebnisse (s. auch Abschnitt 2) zeigen bei allen Arten der deutschen Flora mehr oder weniger starke geographische Muster der genetischen Vielfalt und sprechen daher klar für ein Regiosaatgutkonzept bei Begrünungs- und Renaturierungsmaßnahmen im Sinne der CBD (dritte Ebene der biologischen Vielfalt) und auch des § 40 Bundesnaturschutzgesetz (BNatSchG). Auch wenn sich die teils sehr großzügig bemessenen Ursprungsgebiete nicht immer mit den Mustern genetischer Differenzierung der verwendeten häufigen Pflanzenarten decken (z. B. Listl et al. 2017), reduziert dieses Vorgehen ein unkontrolliertes Einbringen völlig neuer Genotypen in Landschaften und Habitate mit bestehenden Populationen. Für diese pragmatische Herangehensweise ist es wichtig, das Spektrum der Regiosaatgutarten auf weit verbreitete und häufige Arten zu fokussieren. Dieser Grundsatz wird in den Kriterien für die Auswahl von Regiosaatgutarten auch vertreten (Prasse et al. 2010). Arten mit bekannter Aufspaltung in unterschiedliche Sippen – hierzu zählen insbesondere Taxa, die in der Liste der Gefäßpflanzen Deutschlands (Buttler et al. 2018) „Aggregaten“ zugeordnet werden bzw. die sehr starke genetische Differenzierung aufweisen – können somit nur dann als Regiosaatgut vermehrt und eingesetzt werden, wenn die innerartliche genetische Differenzierung gut bekannt ist und mit den
geographisch definierten Ursprungsgebieten übereinstimmt. Dabei ist zu bedenken, dass der derzeitige taxonomische Kenntnisstand für die meisten dieser „Aggregate“ noch auf morphologischen Vergleichen (alpha-taxonomischen Bearbeitungen) und nicht auf Analysen von Artgrenzen mittels evolutionsbiologischer einschließlich molekularer Ansätze beruht. Es ist daher schwer zu sagen, ob die in einem großen Naturraum vertretenen Populationen alle zu derselben biologischen Einheit gehören.
Für die gebotene Stützung und Etablierung neuer Populationen seltener Arten sowie von Arten, die ein hohes Hybridisierungsrisiko aufweisen, muss hingegen aus o. g. Gründen sichergestellt werden, dass dies kontrolliert in behördlich abgestimmten Projektkontexten und nicht wahllos über große Regionen geschieht. Für Arten der Roten Liste, deren Gefährdungssituation zu verringern Ziel der Nationalen Strategie zur biologischen Vielfalt ist (BMU 2007), und hier v. a. für die gefährdeten unter den „Verantwortungsarten“ bietet sich die Ausarbeitung artspezifischer geographischer Managementeinheiten an (vgl. z. B. Duwe et al. 2018; Ismail et al. 2018). Dies kann durch eine enge Kooperation wissenschaftlicher und behördlicher Akteure mit der Naturschutzpraxis erreicht werden.
3.2 Notwendigkeit eines genetischen Monitorings
Der starke Rückgang der Artenvielfalt von Blütenpflanzen in Deutschland ist klar dokumentiert (Wesche et al. 2012; Eichenberg et al. 2020). Die Auswertung der jüngsten Roten Listen zeigt ebenso eine Zunahme der Gefährdung, insbesondere bei Arten nährstoffarmer Standorte und des Grünlands (Metzing et al. 2018). Diese Ergebnisse basieren auf floristischen Daten. Veränderungen in der genetischen Vielfalt innerhalb von Populationen sind demgegenüber deutlich weniger untersucht, obwohl gerade genetische Daten Einblicke in die Qualität der heute vorhandenen Populationen gefährdeter Arten erlauben (Holderegger, Segelbacher 2016). Da bis auf wenige Ausnahmen auch nicht bekannt ist, wie die Populationen innerhalb von Arten genetisch differenziert sind, sind bisher kaum Aussagen dazu
möglich, ob Artenschutzstrategien die genetische Vielfalt repräsentativ berücksichtigen und das evolutive Potenzial von Arten im Blick haben. Weltweit liegen solche Daten bestenfalls für Kulturpflanzen oder wirtschaftlich wichtige Arten vor (Laikre et al. 2010). Das gilt auch für Deutschland und insbesondere für Pflanzen. Für Buche und Fichte wurde ein genetisches Monitoring eingerichtet, um die Anpassungsfähigkeit gegenüber künftigen Umweltveränderungen zu beurteilen. Dazu werden in deutschlandweit verteilten Dauerbeobachtungsflächen neben anderen biologischen Parametern Baumindividuen mit Kernmikrosatelliten untersucht (Schmiedel et al. 2017).
Ein genetisches Monitoring zielt darauf ab, Veränderungen in der genetischen Vielfalt von Arten und die für deren Aufrechterhaltung relevanten Prozesse zu erkennen. Dabei kann es Veränderungen von Allelfrequenzen innerhalb von Populationen genauso geben wie bei der weiträumigeren genetischen Differenzierung, möglicherweise auch durch schnellere Ausbreitung besser angepasster Genotypen im Rahmen des Klimawandels (Anderson, Wadgymar 2020). Eine Herausforderung ist die Erhebung geeigneter Referenzdaten, die v. a. methodisch anschlussfähig sind und auch ein Verständnis der Entstehung bisher vorliegender Diversitätsmuster erlauben. Dies ist durch Kernmikrosatelliten gegeben und wird sich durch weitere Fortschritte in der Entwicklung sequenzbasierter Marker (z. B. single nucleotide polymorphisms, SNP) weiter verbessern (Kasten 1). Auf Grund des Einwanderns von
Neophyten durch den globalen Warenaustausch und des intensiven Einbringens exotischer Arten in die gärtnerische Kultur in Mitteleuropa mit Pollen- und Samenausbreitung in die freie Landschaft wird die Notwendigkeit zur Erkennung artreiner gebietsheimischer Pflanzen in Zukunft eher zunehmen (Bleeker et al. 2007). Hier stellt sich auch die Frage, wie ein Hybridisierungspotenzial rechtzeitig erkannt werden kann. Genetische Überflutung ist eine relevante Gefährdungsursache (Englmaier 2009; Holderegger, Segelbacher 2016) und kann mit molekulargenetischen Methoden zuverlässig detektiert werden.
4 Wie könnte die genetische Forschung für den botanischen Artenschutz organisiert werden?
Naturschutzgenetische Forschung, integrative Taxonomie und DNA-basierte Identifizierung sollten zusammen gedacht werden, denn die Methoden und molekularen Marker haben genauso wie die physischen Objekte und Proben für die Analyse von Artgrenzen und der innerartlichen Vielfalt einen großen Überlappungsbereich. In den meisten Fällen beziehen sich molekulare Daten auf bestimmte Individuen, die in Form von Herbarbelegen nicht nur eine Dokumentation über die Artzuordnung ermöglichen, sondern auch Informationen zu morphologischen Merkmalen und Eigenschaften liefern. Auch wenn bei der Analyse der genetischen Struktur innerhalb einer Population nicht alle beprobten Individuen als Belege konserviert werden können, so lässt sich doch eine Population sicher phylogeographisch einordnen bzw. bei neuen Erkenntnissen über Artgrenzen den aktuell akzeptierten Taxa zuordnen (Kilian et al. 2015). Die Einlagerung entsprechender Gewebe-
oder DNA-Proben ermöglicht es zu jedem Zeitpunkt, von denselben Individuen weitere genetische Daten zu generieren, diese mit den bisherigen Daten zu verknüpfen und dadurch weitere Zusammenhänge aufzuzeigen bzw. Aussagen zu treffen. Dies ist angesichts sich stark entwickelnder genetischer Analyse- und Sequenziermethoden von Bedeutung.
In Deutschland wurden im Rahmen des German-Barcode-of-Life(GBOL)-Projekts (Geiger et al. 2016; gefördert durch das Bundesministerium für Bildung und Forschung, BMBF) Referenzsequenzen für die molekulare Identifikation von Pflanzen generiert, darunter viele Verantwortungsarten. Im Rahmen der Analyse der genetischen Vielfalt gefährdeter Arten (z. B. Ismail et al. 2018, Duwe et al. 2018; gefördert durch das BfN im Rahmen des Umweltforschungsplans) entstanden Synergien bei der Nutzung von Proben und Daten. So lässt sich bei Einsatz der gleichen Genomabschnitte als Barcodes für die Identifizierung von Arten und bei der Analyse innerartlicher Vielfalt sowohl die Artbestimmung als auch die Genauigkeit der Referenzdaten durch Abbildung der innerartlichen Variabilität verifizieren. Sammlungen sind integraler Bestandteil
einer solchen Vernetzung in der Forschung, entsprechend wird z. B. am BGBM in Verbindung mit dem Global Genome Biodiversity Network (Droege et al. 2016) ein DNA-Archiv für die deutsche Flora aufgebaut (Abb. 2), das in Silikagel konservierte Gewebeproben und DNA-Isolate umfasst und damit hohen Qualitätsanforderungen gerecht wird. Auch Herbarien werden durch verbesserte Labormöglichkeiten als Archive zugänglich, in denen Veränderungen der genetischen Vielfalt von Arten erforscht werden können (Rocchetti et al. 2021).
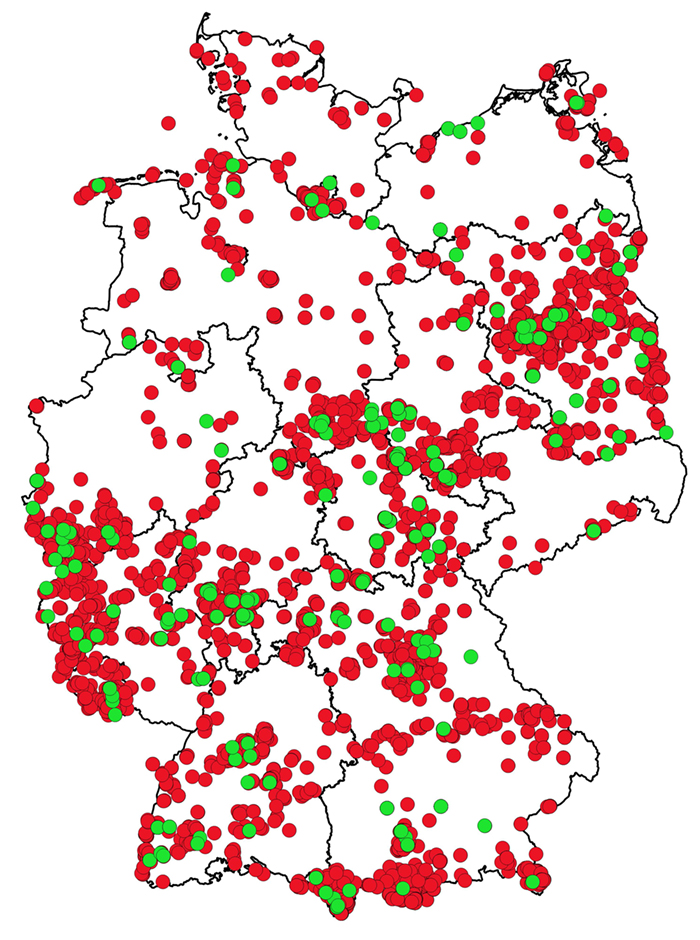
Abb. 2: Repräsentation der Blütenpflanzen aus der deutschen Flora durch Gewebeproben und DNA-Isolate in der DNA-Bank am Botanischen Garten Berlin. Die Karte basiert auf etwa 9 000 Proben von Einzelindividuen oder Populationen, alle durch Herbarbelege dokumentiert (Bild- und Metadaten sind zugänglich über das Virtuelle Herbarium unter http://ww2.bgbm.org/herbarium/default.cfm und das Portal des Global Genome Biodiversity Network unter https://www.ggbn.org/ggbn_portal/). Die grünen Punkte symbolisieren Verantwortungsarten.
Fig. 2: Representation of flowering plants of the German flora in the form of tissue samples and DNA isolates in the DNA Bank of the Berlin Botanical Garden. The map includes some 9,000 samples of individuals or populations, all of which are documented with herbarium specimens (images and metadata available through the virtual herbarium at
http://ww2.bgbm.org/herbarium/default.cfm and the Global Genome Biodiversity Network websites at
https://www.ggbn.org/ggbn_portal/). Green dots represent the “Verantwortungsarten” (= species for whose conservation Germany has a special national responsibility).
Für naturschutzgenetische Fragestellungen gibt es eine Reihe von Methoden (s. Zusammenfassung in Holderegger, Segelbacher 2016). Die überwiegende Zahl der bisher publizierten Studien zur genetischen Vielfalt von Pflanzen in Deutschland basiert auf AFLP (siehe Kasten 1). Durch die Etablierung gut reproduzierbarer Marker, etwa von Kernmikrosatelliten-Loci und SNP, die dann in ergänzenden Beprobungen und Zeitreihen weiterverwendet werden können (Hodel et al. 2016), kann die Anschlussfähigkeit von Datensätzen verbessert werden. Hier kommt es darauf an, dass sich die Wissenschaft für die jeweiligen Arten auf bestimmte Marker einigt.
5 Wie kann der Austausch zwischen Wissenschaft und Praxis verbessert werden?
Die mangelnde Verknüpfung wissenschaftlicher Forschung mit dem Naturschutz ist weltweit ein Problem (Wilson et al. 2016). Populationsgenetische bzw. -genomische Forschung ist ein Paradebeispiel hierfür (Taylor et al. 2017). Mit einer stärkeren Vernetzung von Grundlagenforschung, bedarfs- und anwendungsorientierter Forschung sowie wissenschaftlicher Begleitung von Naturschutzmaßnahmen kann eine gemeinsame Herangehensweise gefördert und erreicht werden. Dies sollte nicht nur auf Ebene einzelner Verbundprojekte stattfinden, wie von Riecken et al. (2020) vorgeschlagen, sondern grundsätzlicher. Bisher werden praktische Artenschutzmaßnahmen im Rahmen von Programmen gefördert, die Forschungskomponenten nicht oder nur in geringem Umfang enthalten dürfen; ein Beispiel ist die Förderung des WIPs-De-Projekts durch das
Bundesprogramm Biologische Vielfalt. Selbst die Planung von Umsetzungsprojekten findet häufig ohne Beteiligung der entsprechenden Stakeholder aus der Wissenschaft statt. Umgekehrt zielt die Planung von Grundlagenforschung, die üblicherweise nicht von Mittelgebern aus dem Naturschutzbereich gefördert wird, häufig auf die Weiterentwicklung von Methoden und das Testen möglichst allgemeingültiger Hypothesen ab, ohne jedoch konkrete Datenbedarfe für Umsetzungsprojekte zu erfüllen. Dabei könnten sich die Grundlagenforschung mit ihren Zielsetzungen und Förderinstrumenten, deren Leistungen nicht am Umsetzungserfolg von Artenschutzmaßnahmen gemessen werden, und die anwendungsorientierte Forschung, die konkret im Artenschutz benötigte Daten und Wissen generiert, inhaltlich aufeinander abstimmen. Dies würde eine breitere Einbeziehung von Stakeholdern im Sinne des Artenschutzes als gesamtgesellschaftliche Aufgabe fördern und sollte auch die Aufbereitung von Forschungsergebnissen
für ein breites Publikum einschließen.
Dabei sollten die Rollen und Möglichkeiten von Institutionen mit naturkundlichen Sammlungen sowie Datenrepositorien (GFBio, NFDI4BioDiversity) mitgedacht werden. Hier ist ein intensiverer Austausch zwischen den unterschiedlichen Akteuren vonnöten, bei dem Erhebung, Strukturierung, Archivierung und Dokumentation sowie Verfügbarmachung von Daten als integraler Bestandteil der Forschungsplanung und -förderung gesehen werden (Holderegger et al. 2019). Dies trägt auch zur Umsetzung der FAIR-Prinzipien (findability, accessibility, interoperability, reuse; https://www.go-fair.org/fair-principles) in Forschung und Forschungsdatenmanagement bei. Mit der Etablierung eines deutschlandweiten Netzwerks, in dem sich Akteure regelmäßig austauschen, und der Schaffung geeigneter Förderinstrumente kann und sollte das gelingen. Auch mit Blick darauf, dass gefährdete
Arten keine Ländergrenzen kennen, ist ein bundesweit abgestimmtes Vorgehen im Artenschutz unabdingbar.
6 Schlussfolgerungen
Angesichts der dramatischen Krise der biologischen Vielfalt auch in Mitteleuropa ist der Dialog zwischen den Akteuren aus naturschutzgenetischer Forschung und Praxis von herausragender Bedeutung. Ein Assessment der genetischen Vielfalt einer Art zeigt phylogeographische Zusammenhänge oder genetische Prozesse wie genetische Drift und genetische Verarmung auf und hilft uns, die Evolutionsgeschichte der betrachteten Art in Raum und Zeit zu verstehen. Genetische Daten können das Management gefährdeter und seltener Arten wirkungsvoll dort unterstützen, wo es um gezielte Eingriffe wie die Schaffung von Biotopverbünden, Populationsstützungen, Wiederansiedlungen oder angesichts des Klimawandels um assisted migration geht (Oostermeijer et al. 2003; Fišer et al. 2021), wo für einen langfristigen und damit nachhaltigen Artenschutz Prioritäten gesetzt werden oder verschiedene
In-situ- und Ex-situ-Schutzmaßnahmen gegeneinander abgewogen werden müssen.
Grundsätzlich schaffen die Globalisierung, der nahezu weltweite Zugang und die damit einhergehende Neuverteilung biologischen Materials eine völlig neue Situation im Vergleich zur traditionellen Landnutzung in regionalen Kontexten mit hauptsächlich regionalem Austausch genetischen Materials. Konkret bedeutet das, dass in einem nie zuvor dagewesenen Ausmaß Saatgut und Pflanzenmaterial unterschiedlichster Genotypen und Arten produziert sowie vermarktet und damit für breite gesellschaftliche Gruppen fast ortsungebunden verfügbar werden. Globale und nationale Strategien zur biologischen Vielfalt haben entsprechend diese Problematik aufgegriffen, z. B. indem sie die Bedeutung der innerartlichen genetischen Vielfalt thematisieren. Auch in der aktuellen wissenschaftlichen Diskussion zur Ausgestaltung und Umsetzung der globalen Ziele biologischer Vielfalt (Post-2020 Global Biodiversity Framework der CBD) wird eine stärkere Berücksichtigung der genetischen Vielfalt
für alle Arten gefordert (Hoban et al. 2020).
In Deutschland gibt es hierzu eine fortschrittliche Gesetzgebung. So fordert § 40 BNatSchG, dass bei der Ausbringung gebietsfremder Arten und damit auch gebietsfremder Genotypen heimischer Arten negative Konsequenzen ausgeschlossen werden müssen. In diesem Zusammenhang ist der Regiosaatgutansatz von großer Bedeutung für die praktische Umsetzung. Allerdings sind die 22 Herkunftsgebiete (Prasse et al. 2010) nicht automatisch anwendbar, da sie nicht bei allen Arten den weiträumigen Mustern der innerartlichen genetischen Differenzierung entsprechen und Aspekte der genetischen Vielfalt innerhalb von Populationen, die für die Sicherung vieler seltener und gefährdeter Arten entscheidend sind, gar nicht berücksichtigen können. Seltene und gefährdete Arten (hier geben die Roten Listen pragmatisch verwendbare Informationen) sollten daher aus der Vermehrung und Vermarktung als Regiosaatgut herausgenommen werden.
Für diese Arten bietet sich eine Umsetzung von Erhaltungsstrategien in Naturschutzprojekten und Programmen an (z. B. WIPs-De; Zippel et al. 2016), im Rahmen derer nicht nur die Beachtung wissenschaftlicher Standards gewährleistet werden kann, sondern auch eine breite gesellschaftliche Einbeziehung von Akteuren des Naturschutzes. Es bedarf außerdem einer konsequenten Anwendung des Artenfilterprinzips (https://regionalisierte-pflanzenproduktion.de/artenfilter). Hierzu ist es erforderlich, komplexe Artengruppen mit oft unklaren Artgrenzen spezifischer zu untersuchen (mittels integrativer Taxonomie unter Einbeziehung evolutionsbiologischer und molekulargenetischer Methoden), um zuverlässige Daten für die Anwendung eines Artenfilters zu erlangen.
Von entscheidender Bedeutung ist ein wissensbasiertes Management der Artenvielfalt. Arten müssen dabei als biologische Einheiten (nicht Morphospezies) verstanden werden. Dazu gehört das Verständnis reproduktiver Mechanismen, da die Genpools von Pflanzenarten über den Austausch von Pollen und Diasporen beeinflusst werden. Es gilt, lebendige Strukturen für die Interaktion zwischen Forschungseinrichtungen mit entsprechenden Laborkapazitäten und methodischer Expertise sowie den Akteuren des Naturschutzes in Behörden und Verbänden zu schaffen. Nur so können Fragestellungen aus der Praxis zeitnah Eingang in wissenschaftliche Untersuchungen finden und unter enger Zusammenarbeit zwischen praktischen Maßnahmen zur Sicherung gefährdeter Arten und genetischer Forschung in konkrete Handlungsempfehlungen münden.
7 Literatur
↑
Aguilar R., Quesada M. et al. (2008): Genetic consequences of habitat fragmentation in plant populations: Susceptible signals in plant traits and methodological approaches. Molecular Ecology 17(24): 5 177 – 5 188. DOI: 10.1111/j.1365-294X.2008.03971.x
↑
Albach D., Schönswetter P., Tribsch A. (2006): Comparative phylogeography of the Veronica alpina complex in Europe and North America. Molecular Ecology 15(11): 3 269 – 3 286. DOI: 10.1111/j.1365-294X.2006.02980.x
↑
Anderson J.T., Wadgymar S.M. (2020): Climate change disrupts local adaptation and favors upslope migration. Ecology Letters 23(1): 181 – 192. DOI: 10.1111/ele.13427
↑
Avise J.C. (2000): Phylogeography: the history and formation of species. Harvard University Press. Cambridge, MA: 464 S.
↑
Barrett S.C. (2010): Understanding plant reproductive diversity. Philosophical Transactions Royal Society B 365(1 537): 99 – 109. DOI: 10.1098/rstb.2009.0199
↑
Becker T. (2003): Auswirkungen langzeitiger Fragmentierung auf Populationen am Beispiel der reliktischen Steppenrasenart Astragalus exscapus L. (Fabaceae). Dissertationes Botanicae 380: 210 S.
↑
Betz C., Scheuerer M., Reisch C. (2013): Population reinforcement – A glimmer of hope for the conservation of the highly endangered Spring Pasque flower (Pulsatilla vernalis). Biological Conservation 168: 161 – 167. DOI: 10.1016/j.biocon.2013.10.004
↑
Bleeker W., Schmitz U., Ristow M. (2007): Interspecific hybridization between alien and native plant species in Germany and its consequences for native biodiversity. Biological Conservation 137(2): 248 – 253. DOI: 10.1016/j.biocon.2007.02.004
↑
BMU/Bundesministerium für Umwelt, Naturschutz und Reaktorsicherheit (Hrsg.) (2007): Nationale Strategie zur biologischen Vielfalt. BMU. Berlin: 178 S.
↑
Borsch T., Berendsohn W.G. et al. (2020): World Flora Online: Placing taxonomists at the heart of a definitive and comprehensive global resource on the world's plants. Taxon 69(6): 1 311 – 1 341. DOI: 10.1002/tax.12373
↑
Buttler K.P., May R., Metzing D. (2018): Liste der Gefäßpflanzen Deutschlands. Florensynopse und Synonyme. BfN-Skripten 519: 286 S.
↑
Comes H.P., Kadereit J.W. (1998): The effect of Quarternary climatic changes on plant distribution and evolution. Trends in Plant Science 3(11): 432 – 438. DOI: 10.1016/S1360-1385(98)01327-2
↑
Droege G., Barker, K. et al. (2016): The Global Genome Biodiversity Network (GGBN) data standard specification. Database: baw125. DOI: 10.1093/database/baw125
↑
Durka W., Michalski S.G. et al. (2017): Genetic differentiation within multiple common grassland plants supports seed transfer zones for ecological restoration. Journal of Applied Ecology 54(1): 116 – 126. DOI: 10.1111/1365-2664.12636
↑
Durka W., Nosslo C. et al. (2013): Extreme genetic depauperation and differentiation of both populations and species in Eurasian feather grasses (Stipa). Plant Systematics and Evolution 299(1): 259 – 299. DOI: 10.1007/s00606-012-0719-0
↑
Duwe V.K., Ismail S.A. et al. (2015): Fourteen polymorphic microsatellite markers for the threatened Arnica montana (Asteraceae). Applications in Plant Sciences 3(1): 1 400 091. DOI:10.3732/apps.1400091
↑
Duwe V.K., Muller L.A. et al. (2016): Development of microsatellite markers for Crepis mollis (Asteraceae). Applications in Plant Sciences 4(7): 1 600 022. DOI: 10.3732/apps.1600022
↑
Duwe V.K., Muller L.A. et al. (2017): Pervasive genetic differentiation among Central European populations of the threatened Arnica montana L. and genetic erosion at lower elevations. Perspectives in Plant Ecology, Evolution and Systematics 27: 45 – 56. DOI: 10.1016/j.ppees.2017.02.003
↑
Duwe V.K., Muller L.A. et al. (2018): Genetic structure and genetic diversity of the endangered grassland plant Crepis mollis (Jacq.) Asch. as a basis for conservation management in Germany. Conservation Genetics 19(3): 527 – 543. DOI: 10.1007/s10592-017-1025-8
↑
Eichenberg D., Bowler D.E. et al. (2020): Widespread decline in Central European plant diversity across six decades. Global Change Biology 27(5): 1 097 – 1 110. DOI: 10.1111/gcb.15447
↑
Eimert K., Hüwe U. et al. (2016): Evaluation of genetic differentiation of autochthonous sloe (Prunus spinosa, Rosaceae) populations across Germany using molecular markers. Plant Ecology and Evolution 149(3): 280 – 290. DOI: 10.5091/plecevo.2016.1182
↑
Engelhardt D.E., Lloyd M.W., Neel M.C. (2014): Effects of genetic diversity of conservation and restoration potential at individual, population and regional scales. Biological Conservation 179: 6 – 16. DOI: 10.1016/j.biocon.2014.08.011
↑
Englmaier P. (2009): Die Ansaat borstblättriger Schwingelarten (Festuca spp.): Naturnahe Begrünung oder Florenverfälschung? Gredleriana 9: 61 – 82.
↑
Fišer Ž., Aronne G. et al. (2021): ConservePlants: An integrated approach to conservation of threatened plants for the 21st Century. Research Ideas and Outcomes 7: e62810. DOI: 10.3897/rio.7.e62810
↑
Geiger M.F., Astrin J.J. et al. (2016): How to tackle the molecular species inventory for an industrialized nation – lessons from the first phase of the German Barcode of Life initiative GBOL. Genome 59(9): 1 – 10. DOI: 10.1139/gen-2015-0185
↑
Godefroid S., Piazza C. et al. (2011): How successful are plant species reintroductions? Biological Conservation 144(2): 672 – 682. DOI: 10.1016/j.biocon.2010.10.003
↑
Grivet D., Sork V.L. et al. (2008): Conserving the evolutionary potential of California valley oak (Quercus lobata Née): a multivariate genetic approach to conservation planning. Molecular Ecology 17(1): 139 – 156. DOI: 10.1111/j.1365-294X.2007.03498.x
↑
Gruttke H., Ludwig G. et al. (2004): Memorandum: Verantwortlichkeit Deutschlands für die weltweite Erhaltung von Arten – verabschiedet durch das Symposium: „Ermittlung der Verantwortlichkeit für die weltweite Erhaltung von Tierarten mit Vorkommen in Mitteleuropa“, Vilm, 17. – 20. November 2003. Naturschutz und Biologische Vielfalt 8: 273 – 280.
↑
Harter D.E., Jentsch A., Durka W. (2015): Holocene re-colonisation, central-marginal distribution and habitat specialisation shape population genetic patterns within an Atlantic European grass species. Plant Biology 17(3): 684 – 693. DOI: 10.1111/plb.12269
↑
Hensen I., Kilian C. et al. (2010): Low genetic variability and strong differentiation among isolated populations of the rare steppe grass Stipa capillata L. in central Europe. Plant Biology 12(3): 526 – 536. DOI: 10.1111/j.1438-8677.2009.00227.x
↑
Hoban S., Bruford M. et al. (2020): Genetic diversity targets and indicators in the CBD post-2020 Global Biodiversity Framework must be improved. Biological Conservation 248: 108 654. DOI: 10.1016/j-biocon.2020.108654
↑
Hodel R.G., Segovia-Salcedo C. et al. (2016): The report of my death was an exaggeration: A review for researchers using microsatellites in the 21st century. Applications in Plant Sciences 4(6): 1 600 025. DOI: 10.3732/apps.160025
↑
Holderegger R., Balkenhol N. et al. (2019): Conservation genetics: Linking science with practice. Molecular Ecology 28(17): 3 848 – 3 856. DOI: 10.1111/mec.15202
↑
Holderegger R., Segelbacher G. (Hrsg.) (2016): Naturschutzgenetik. Ein Handbuch für die Praxis. Haupt. Bern: 247 S.
↑
Höltken A.M., Hennig A. et al. (2017): Erhaltung und Produktion gebietseigener genetischer Vielfalt in Ex-situ-Populationen. Umsetzung der Ergebnisse aus DNA-Studien am Wildapfel. Naturschutz und Landschaftsplanung 49(4): 126 – 134.
↑
Huck S., Büdel B., Schmitt T. (2012): Ice-age isolation, postglacial hybridization and recent population bottlenecks shape the genetic structure of Meum athamanticum in Central Europe. Flora 207(6): 399 – 407. DOI: 10.1016/j.flora.2012.03.005
↑
Ismail S.A., Duwe V.K. et al. (2015): Development of ten polymorphic microsatellite markers for Scabiosa canescens Waldst. & Kit. (Dipsacaceae). Conservation Genetics Resources 7(1): 725 – 728. DOI: 10.1007/s12686-015-0446-2
↑
Ismail S.A., Duwe V.K. et al. (2018): Assessment of current genetic structure from local to geographic scales indicates brake down of historically extensive gene flow in the dry grassland species Scabiosa canescens Waldst. & Kit. (Dipsacaceae). Diversity and Distributions 24(2): 233 – 243. DOI: 10.1111/ddi/12667
↑
Jelić M., Patenković A. et al. (2015): Indigenous forests of European black poplar along the Danube River: Genetic structure and reliable detection of introgression. Tree Genetics and Genomes 11(5): 89. DOI: 10.1007/s11295-015-0915-5
↑
Kaulfuß F., Reisch C. (2017): Reintroduction of the endangered and endemic plant species Cochlearia bavarica – Implications from conservation genetics. Ecology and Evolution 7(24): 1 100 – 1 112. DOI: 10.1002/ece3.3596
↑
Keller M., Kollmann J., Edwards P.J. (2000): Genetic introgression from distant provenances reduces fitness in local weed populations. Journal of Applied Ecology 37(4): 647 – 659. DOI: 10.1046/j.1365-2664.2000.00517.x
↑
Kilian N., Henning T. et al. (2015): Sample data processing in an additive and reproducible taxonomic workflow by using character data persistently linked to preserved individual specimens. Database 2015: bav094. DOI:10.1093/database/bav094
↑
Kropf M., Bardy K. et al. (2020): Phylogeographical structure and genetic diversity of Adonis vernalis L. (Ranunculaceae) across and beyond the Pannonian region. Flora 262: 151497.
↑
Laikre L., Fred W. et al. (2010): Neglect of genetic diversity in implementation of the convention on biological diversity. Conservation Biology 24(1): 86 – 88. DOI: 10.1111/j.1523-1739.2009.01425.x
↑
Lauterbach D., Burkart M. et al. (2019): Beiträge der Botanischen Gärten Potsdam und Berlin zum botanischen Artenschutz in Brandenburg. Naturschutz und Landschaftspflege in Brandenburg 28(1): 4 – 19.
↑
Leimu R., Fischer M. (2008): A meta-analysis of local adaptation in plants. PLOS ONE 3(12): e4010. DOI: 10.1371/journal.pone.0004010
↑
Leipold M., Tausch S. et al. (2017): Species distribution modeling and molecular markers suggest longitudinal range shifts and cryptic northern refugia of the typical calcareous grassland species Hippocrepis comosa (horseshoe vetch). Ecology and Evolution 7(6): 1 – 17. DOI: 10.1002/ece3.2811
↑
Lesica P., Allendorf F.W. (1995): When are peripheral populations valuable for conservation? Conservation Biology 9(4): 753 – 760. DOI: 10.1046/j.1523-1739.1995.09040753.x
↑
Listl D., Poschlod P., Reisch C. (2017): Do seed transfer zones for ecological restoration reflect the spatial genetic variation of the common grassland species Lathyrus pratensis? Restoration Ecology 26(4): 667 – 676. DOI: 10.1111/rec.12613
↑
Ludwig G., May R., Otto C. (2007): Verantwortlichkeit Deutschlands für die weltweite Erhaltung der Farn- und Blütenpflanzen – vorläufige Liste. BfN-Skripten 220: 32 S. + Anhang.
↑
Meindl C., Brune V. et al. (2016): Survival and postglacial immigration of the steppe plant Scorzonera purpurea to Central Europe. Plant Systematics and Evolution 302(8): 971 – 984. DOI: 10.1007/s00606-016-1311-9
↑
Metzing D., Garve E. et al. (2018): Rote Liste und Gesamtartenliste der Farn- und Blütenpflanzen (Trachaeophyta) Deutschlands. Naturschutz und Biologische Vielfalt 70(7): 13 – 358.
↑
Mijangos J.L., Pacioni C. et al. (2014): Contribution of genetics to ecological restoration. Molecular Ecology 24(1): 22 – 37.
↑
Montalvo A.M., Ellstrand N.C. (2001): Nonlocal transplantation and outbreeding depression in the subshrub Lotus scoparius (Fabaceae). American Journal of Botany 88(2): 258–269. DOI: 10.2307/2657017
↑
Naciri Y., Linder H.P. (2015): Species delimitation and relationships: The dance of the seven veils. Taxon 64(1): 3–16. DOI: 10.12705/641.24
↑
NetPhyD, BfN/Netzwerk Phytodiversität Deutschlands, Bundesamt für Naturschutz (Hrsg.) (2013): Verbreitungsatlas der Farn- und Blütenpflanzen Deutschlands. Landwirtschaftsverlag. Münster: 912 S.
↑
Nierbauer K.U., Kanz B., Zizka G. (2014): The widespread naturalisation of Nymphaea hybrids is masking the decline of wild-type Nymphaea alba in Hesse, Germany. Flora 209(2): 122–130. DOI: 10.1016/j.flora.2013.12.005
↑
Oostermeijer J.G., Luijten S.H., den Nijs J.C. (2003): Integrating demographic and genetic approaches in plant conservation. Biological Conservation 113(3): 389–398. DOI: 10.1016/S0006-3207(03)00127-7
↑
Pagel E., Lehmair T.A. etal. (2020): Genetic variation of typical plant species in hay meadows: The effect of land use history, landscape structure, and habitat quality. Frontiers in Ecology and Evolution 8: e593302. DOI: 10.3389/fevo.2020.593302
↑
Poschlod P. (2015): Geschichte der Kulturlandschaft. Ulmer. Stuttgart: 320S.
↑
Prasse R., Kunzmann D., Schröder R. (2010): Entwicklung und praktische Umsetzung naturschutzfachlicher Mindestanforderungen an einen Herkunftsnachweis für gebietseigenes Wildpflanzensaatgut krautiger Pflanzen. Abschlussbericht. Deutsche Bundesstiftung Umwelt. Hannover: 166 S.
↑
Putz C.M., Schmid C., Reisch C. (2015): Living in isolation – population structure, reproduction, and genetic variation of the endangered plant species Dianthus gratianopolitanus (Cheddar pink). Ecology and Evolution 5(17): 3 610 – 3 621. DOI: 10.1002/ece3.1611
↑
Reichel K., Richter F. et al. (2016): Genetic diversity in the locally declining Laserpitium prutenicum L. and the more common Selinum carvifolia (L.) L.: A “silent goodbye”? Conservation Genetics 17(4): 847 – 860. DOI: 10.1007/s10592-016-0827-4
↑
Reim S., Lochschmidt F. et al. (2017): Species delimitation, genetic diversity and structure of the European indigenous wild pear (Pyrus pyraster) in Saxony, Germany. Genetic Resources and Crop Evolution 64(5): 1 075 – 1 085. DOI: 10.1007/s10722-016-0426-8
↑
Reisch C., Rosbakh S. (2021): Patterns of genetic variation in European plant species depend on altitude. Diversity and Distributions 27(1): 157 – 163. DOI: 10.1111/ddi.13177
↑
Riecken U., Ammer C. et al. (2020): Notwendigkeit eines Brückenschlags zwischen Wissenschaft und Praxis im Naturschutz – Chancen und Herausforderungen. Natur und Landschaft 95(8): 364 – 371. DOI: 10.17433/8.2020.50153829.364-371
↑
Rieseberg L.H., Willis J.H. (2007): Plant speciation. Science 317(5 840): 910 – 914. DOI: 10.1126/science.1137729
↑
Rocchetti G.A., Armstrong C.G. et al. (2021): Reversing extinction trends: New uses of (old) herbarium specimens to accelerate conservation action on threatened species. New Phytologist 230(2): 433 – 450. DOI: 10.1111/nph.17133
↑
Schmiedel J., Tröber U. et al. (2017): GenMon – Implementation of a genetic monitoring system in European Beech (Fagus sylvatica L.) and Norway Spruce (Picea abies [L.] Karst) populations in Germany. Thünen Report 62: 76 – 81. DOI: 10.3220/REP1539855736000
↑
Shafer A.B., Wolf J.B. et al. (2014): Genomics and the challenging translation into conservation practice. Trends in Ecology and Evolution 30(2): 78 – 87. DOI: 10.1016/j.tree.2014.11.009
↑
Taberlet P., Fumagalli L. et al. (1998): Comparative phylogeography and postglacial colonization routes in Europe. Molecular Ecology 7(4): 453 – 464. DOI: 10.1046/j.1365-294x.1998.00289.x
↑
Taylor H.R., Dussex N., van Heezik Y. (2017): Bridging the conservation genetics gap by identifying barriers to implementation for conservation practitioners. Global Ecology and Conservation 10: 231 – 242. DOI: 10.1016/j.gecco.2017.04.001
↑
Titze A., Hepting C. et al. (2020): Wilde Arnika – ein Leitfaden für die Praxis. ArnikaHessen. Botanischer Garten der Phillips-Universität Marburg. Marburg: 229 S.
↑
Van Geert A., van Rossum F., Triest L. (2008): Genetic diversity in adult and seedling populations of Primula vulgaris in a fragmented agricultural landscape. Conservation Genetics 9(4): 845 – 853. DOI: 10.1007/s10592-007-9409-9
↑
Van Rossum F., Hardy O.J. et al. (2020): Genetic monitoring of translocated plant populations in practice. Molecular Ecology 29(21): 4 040 – 4 058. DOI: 10.1111/mec.15550
↑
Weiss-Schneeweiss H., Emadzade K. et al. (2013): Evolutionary consequences, constraints and potential of polyploidy in plants. Cytogenetic and Genome Research 140(2 – 4): 137 – 150. DOI: 10.1159/000351727
↑
Welk E. (2002): Arealkundliche Analyse und Bewertung der Schutzrelevanz seltener und gefährdeter Gefäßpflanzen Deutschlands. Schriftenreihe für Vegetationskunde 37: 21 – 187.
↑
Wesche K., Krause B. et al. (2012): Fifty years of change in Central European grassland vegetation: Large losses in species richness and animal-pollinated plants. Biological Conservation 150(1): 76 – 85. DOI: 10.1016/j.biocon.2012.02.015
↑
Wilson K., Auerbach N. et al. (2016): Conservation research is not happening where it is most needed. PLOS Biology 14(3): e1002413. DOI: 10.1371/journal.pbio.1002413
↑
Zippel E., Borgmann P. et al. (2016): Verknüpfung von ex situ und in situ-Artenschutz: das Projekt „Wildpflanzenschutz in Deutschland“. In: Korn H., Bockmühl K., Schliep R. (Hrsg.): Biodiversität und Klima – Vernetzung der Akteure in Deutschland XII. BfN-Skripten 432: 49 – 57.
Förderung und Dank
Wir danken dem Bundesamt für Naturschutz (BfN) für die Förderung des Projekts „Integration von Ex-situ- und In-situ-Maßnahmen zur Erhaltung gefährdeter Blütenpflanzen in Deutschland – ein Modellvorhaben zur Umsetzung der Global Strategy for Plant Conservation (GSPC)“, FKZ 3512-86-0400, mit Mitteln des Bundesumweltministeriums (BMU), dem Verein der Freunde des BGBM e. V. und dem Jutta-Krieg-Forschungsfonds am BGBM für weitere wesentliche Unterstützung. Gabriele Dröge (Berlin) danken wir für die Generierung der Karte für Abb. 2.



